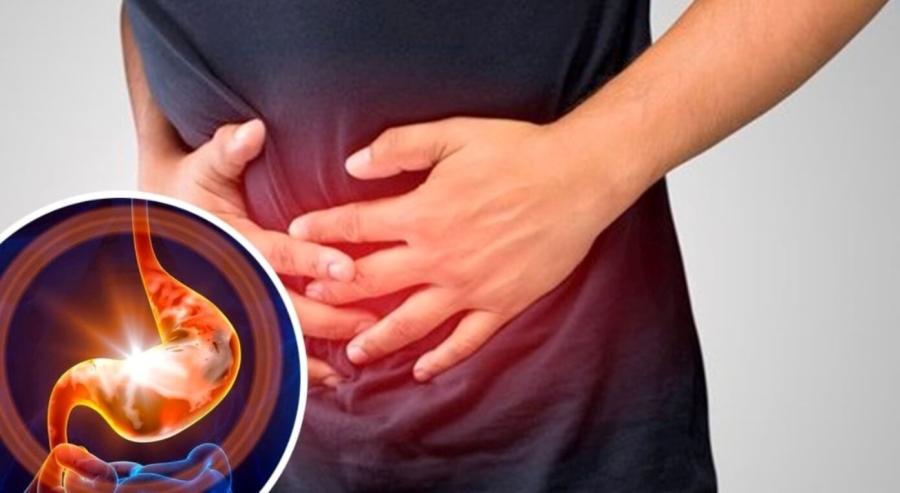
Спровоцировать воспаление могут инфекционные заболевания и аутоиммунные процессы. У больных наблюдается волнообразное повышение температуры, вялость, усталость, режущая боль в животе, тошнота, рвота, понос, вздутие живота, потеря веса. Кожные покровы становятся сухими и тусклыми, волосы выпадают.
Питание при болезни Крона

Болезнь Крона – это хроническое заболевание пищеварительного тракта, сопровождающаяся воспалительной реакцией. Для недуга характерно появление гранулем и изменением структуры эпителия, который выстилает внутреннюю поверхность кишечника.
Болезнь Крона может поражать все отделы ЖКТ, начиная от ротовой полости и заканчивая анальным отверстием, но чаще всего патология затрагивает подвздошную кишку. Это неизлечимое заболевание и порой рецидив появляется по прошествии двадцати лет. Большую роль в формировании недуга играет генетическая предрасположенность.
Спровоцировать воспаление могут инфекционные заболевания и аутоиммунные процессы. У больных наблюдается волнообразное повышение температуры, вялость, усталость, режущая боль в животе, тошнота, рвота, понос, вздутие живота, потеря веса. Кожные покровы становятся сухими и тусклыми, волосы выпадают.
Болезнь Крона является довольно серьезным недугом, который способен привести к внутреннему кровотечению, язвенной болезни, перитониту, а также образованию свищей и абсцессов. При любых заболеваниях пищеварительного тракта важно соблюдать диетическое питание, и болезнь Крона не является исключением.
Правильное питание помогает избежать рецидива и продлить период ремиссии. Диета при болезни Крона является основным элементом лечебных мероприятий. Она поможет восстановить прежнюю работу пищеварительной системы и обогатить организм витаминами и питательными веществами.
Меню составляется лечащим врачом или диетологом исключительно для каждого пациента. Оно должно быть составлено таким образом, чтобы пища смогла проникнуть через воспаленные участки и не навредить органу еще больше. Выбирая продукты для приготовления нужно в первую очередь думать не о вкусе, а о пользе. В приоритете должно стоять ваше здоровье.
Строгость диеты при болезни Крона напрямую зависит от степени тяжести патологического процесса. Если недуг затронул только лишь подвздошную кишку, то ограничения будут не слишком строгими, а если в процесс вовлекается желудок и двенадцатиперстная кишка, тогда придется придерживаться строгой диеты.
Рассыпчатые каши в меню постепенно заменяют перетертыми блюдами. Для поддержания всего организма увеличивается употребление продуктов с большим содержанием калия и кальция. Вводятся разные разрешенные продукты для того, чтобы разнообразить рацион и обогатить организм необходимыми витаминами и минералами.
Чем помогает диета №4?
Болезнь Крона представляет собой воспалительный процесс, приводящий к изменению внутренней стенки кишечника. Чаще всего заболевание локализуется в тонкой или толстой кишке. Способ лечения, дающий шанс навсегда распрощаться с болезнью Крона, до сих пор не найден, так как причина ее появления остается до конца неизученной.
Комплексная терапия в сочетании медикаментов и строгой диеты направлена лишь на отсрочку очередного обострения. Питаться, соблюдая предписанные врачом правила, значит, помогать своему организму находиться в состоянии стойкой ремиссии.
Специальная диета при болезни Крона не представлена медициной. Из разработанных лечебных способов питания наиболее подходящей считается стол №4. Главной задачей является исключение продуктов, провоцирующих газообразование в желудочно-кишечном тракте. Поступающие продукты не должны раздражать слизистую физически, химически или термически. В результате этого будет достигнуто:
- уменьшение нагрузки на весь желудочно-кишечный тракт, включая толстую кишку;
- создание условий оздоровления слизистой оболочки и восстановления разрушенных тканей;
- замедление перистальтики кишечника и нормализация стула.
Больному необходимо понять, что соблюдение диеты при болезни Крона является частью лечебного процесса. Малейшее нарушение, особенно во время обострения, может привести к ухудшению и повлечь за собой такие последствия, как перфорация кишечника или его непроходимость. Чтобы улучшить состояние организма, важно строго придерживаться требований диеты при Кроне и не отступать от них.
Метаанализ педиатрических исследований показал, что частота ремиссии с EEN была эквивалентна таковой для кортикостероидов [118], а другие исследования показали более высокую частоту ремиссии при БК подвздошной кишки или подвздошно-толстокишечной БК, чем при толстокишечном фенотипе [119]. [119]. Из-за снижения вкусовых качеств EEN, в клинической практике пища обычно медленно вводится после 8–12 недель EEN. Во многих случаях это приводит к рецидиву заболевания, однако период ремиссии позволяет инициировать иммуномодуляторы, которые могут занять недели, чтобы стать эффективными. Поскольку считается, что дисбактериоз кишечника играет роль в патогенезе ВЗК, трансплантация фекальной микробиоты (FMT) является эффективной стратегией восстановления кишечного микробного разнообразия и, как сообщается, имеет потенциальную терапевтическую ценность при ВЗК. В недавнем исследовании сообщалось о комплексной интегративной терапии под названием «усиленная стратегия FMT», которая была полезна при лечении стероид-зависимых пациентов с ВЗК. Эта стратегия состоит из запланированных FMTs в сочетании со стероидами, лечением антителами против TNF-α или энтеральным питанием [120].
Литература:
- Eckmann, L. Animal models of inflammatory bowel disease: Lessons from enteric infections. Ann. N. Y. Acad. Sci.2006, 1072, 28–38. [Google Scholar] [CrossRef] [PubMed]
- Yan, F.; Wang, L.; Shi, Y.; Cao, H.; Liu, L.; Washington, M.K.; Chaturvedi, R.; Israel, D.A.; Cao, H.; Wang, B.; et al. Berberine promotes recovery of colitis and inhibits inflammatory responses in colonic macrophages and epithelial cells in DSS-treated mice. Am. J. Physiol. Gastrointest. Liver Physiol.2012, 302, G504–G514. [Google Scholar] [CrossRef] [PubMed]
- Baumgart, D.C.; Sandborn, W.J. Crohn’s disease. Lancet2012, 380, 1590–1605. [Google Scholar] [CrossRef]
- Ordas, I.; Eckmann, L.; Talamini, M.; Baumgart, D.C.; Sandborn, W.J. Ulcerative colitis. Lancet2012, 380, 1606–1619. [Google Scholar] [CrossRef]
- Ferguson, L.R.; Peterman, I.; Hubner, C.; Philpott, M.; Shellin, A.N. Uncoupling gene-diet in inflammatory bowel disease (IBD). Genes Nutr.2007, 2, 71–73. [Google Scholar] [CrossRef] [PubMed]
- Reiff, C.; Kelly, D. Inflammatory bowel disease, gut bacteria and probiotic therapy. Int. J. Med. Microbiol.2010, 300, 25–33. [Google Scholar] [CrossRef] [PubMed]
- Molodecky, N.A.; Soon, I.S.; Rabi, D.M.; Ghali, W.A.; Ferris, M.; Chernoff, G.; Benchimol, E.I.; Panaccione, R.; Ghosh, S.; Barkema, H.W.; et al. Increasing incidence and prevalence of the inflammatory bowel diseases with time, based on systematic review. Gastroenterology2012, 142, 46–54. [Google Scholar] [CrossRef] [PubMed]
- Ng, S.C.; Bernstein, C.N.; Vatn, M.H.; Lakatos, P.L.; Loftus, E.V., Jr.; Tysk, C.; O’Morain, C.; Moum, B.; Colombel, J.F. Geographical variability and environmental risk factors in inflammatory bowel disease. Gut2013, 62, 630–649. [Google Scholar] [CrossRef] [PubMed]
- Cosnes, J.; Gower-Rousseau, C.; Seksik, P.; Cortot, A. Epidemiology and natural history of inflammatory bowel diseases. Gastroenterology2011, 140, 1785–1794. [Google Scholar] [CrossRef] [PubMed]
- Burisch, J.; Munkholm, P. Inflammatory bowel disease epidemiology. Curr. Opin. Gastroenterol.2013, 29, 357–362. [Google Scholar] [CrossRef] [PubMed]
- Kappelman, M.D.; Rifas-Shiman, S.L.; Kleinman, K.; Ollendorf, D.; Bousvaros, A.; Grand, R.J.; Finkelstein, J.A. The prevalence and geographic distribution of Crohn’s disease and ulcerative colitis in the USA. Clin. Gastroenterol. Hepatol.2007, 5, 1424–1429. [Google Scholar] [CrossRef] [PubMed]
- Karolewska-Bochenek, K.; Lazowska-Przeorek, I.; Albrecht, P.; Grzybowska, K.; Ryzko, J.; Szamotulska, K.; Radzikowski, A.; Landowski, P.; Krzesiek, E.; Ignys, I.; et al. Epidemiology of inflammatory bowel disease among children in poland. A prospective, population-based, 2-year study, 2002–2004. Digestion2009, 79, 121–129. [Google Scholar] [CrossRef] [PubMed]
- Ward, L.M.; Rauch, F.; Matzinger, M.A.; Benchimol, E.I.; Boland, M.; Mack, D.R. Iliac bone histomorphometry in children with newly diagnosed inflammatory bowel disease. Osteoporos. Int.2010, 21, 331–337. [Google Scholar] [CrossRef] [PubMed]
- Thia, K.T.; Loftus, E.V., Jr.; Sandborn, W.J.; Yang, S.K. An update on the epidemiology of inflammatory bowel disease in Asia. Am. J. Gastroenterol.2008, 103, 3167–3182. [Google Scholar] [CrossRef] [PubMed]
- Hommes, D.; Colombel, J.F.; Emery, P.; Greco, M.; Sandborn, W.J. Changing Crohn’s disease management: Need for new goals and indices to prevent disability and improve quality of life. J. Crohns Colitis2012, 6, S224–S234. [Google Scholar] [CrossRef]
- Kappelman, M.D.; Palmer, L.; Boyle, B.M.; Rubin, D.T. Quality of care in inflammatory bowel disease: A review and discussion. Inflamm. Bowel Dis.2010, 16, 125–133. [Google Scholar] [CrossRef] [PubMed]
- Gibson, T.B.; Ng, E.; Ozminkowski, R.J.; Wang, S.; Burton, W.N.; Goetzel, R.Z.; Maclean, R. The direct and indirect cost burden of Crohn’s disease and ulcerative colitis. J. Occup. Environ. Med.2008, 50, 1261–1272. [Google Scholar] [CrossRef] [PubMed]
- Okou, D.T.; Kugathasan, S. Role of genetics in pediatric inflammatory bowel disease. Inflamm. Bowel Dis.2014, 20, 1878–1884. [Google Scholar] [CrossRef] [PubMed]
- Franke, A.; McGovern, D.P.; Barrett, J.C.; Wang, K.; Radford-Smith, G.L.; Ahmad, T.; Lees, C.W.; Balschun, T.; Lee, J.; Roberts, R.; et al. Genome-wide meta-analysis increases to 71 the number of confirmed Crohn’s disease susceptibility loci. Nat. Genet.2010, 42, 1118–1125. [Google Scholar] [CrossRef] [PubMed]
- Fiocchi, C. Genes and ”in-vironment”: How will our concepts on the pathophysiology of inflammatory bowel disease develop in the future? Dig. Dis.2012, 30, 2–11. [Google Scholar] [CrossRef] [PubMed]
- Chassaing, B.; Darfeuille-Michaud, A. The commensal microbiota and enteropathogens in the pathogenesis of inflammatory bowel diseases. Gastroenterology2011, 140, 1720–1728. [Google Scholar] [CrossRef] [PubMed]
- Macdonald, T.T. New cytokine targets in inflammatory bowel disease. Gastroenterol Hepatol2011, 7, 474–476. [Google Scholar]
- Cho, J.H.; Abraham, C. Inflammatory bowel disease genetics: Nod2. Annu. Rev. Med.2007, 58, 401–416. [Google Scholar] [CrossRef] [PubMed]
- Orholm, M.; Binder, V.; Sorensen, T.I.; Rasmussen, L.P.; Kyvik, K.O. Concordance of inflammatory bowel disease among Danish twins. Results of a nationwide study. Scand. J. Gastroenterol.2000, 35, 1075–1081. [Google Scholar] [PubMed]
- Halme, L.; Paavola-Sakki, P.; Turunen, U.; Lappalainen, M.; Farkkila, M.; Kontula, K. Family and twin studies in inflammatory bowel disease. World J. Gastroenterol.2006, 12, 3668–3672. [Google Scholar] [CrossRef] [PubMed]
- Tysk, C.; Lindberg, E.; Jarnerot, G.; Floderus-Myrhed, B. Ulcerative colitis and Crohn’s disease in an unselected population of monozygotic and dizygotic twins. A study of heritability and the influence of smoking. Gut1988, 29, 990–996. [Google Scholar] [CrossRef] [PubMed]
- Yang, H.; Rotter, J.I.; Toyoda, H.; Landers, C.; Tyran, D.; McElree, C.K.; Targan, S.R. Ulcerative colitis: A genetically heterogeneous disorder defined by genetic (HLA class II) and subclinical (antineutrophil cytoplasmic antibodies) markers. J. Clin. Invest.1993, 92, 1080–1084. [Google Scholar] [CrossRef] [PubMed]
- Barrett, J.C.; Hansoul, S.; Nicolae, D.L.; Cho, J.H.; Duerr, R.H.; Rioux, J.D.; Brant, S.R.; Silverberg, M.S.; Taylor, K.D.; Barmada, M.M.; et al. Genome-wide association defines more than 30 distinct susceptibility loci for Crohn’s disease. Nat. Genet.2008, 40, 955–962. [Google Scholar] [CrossRef] [PubMed]
- Anderson, C.A.; Boucher, G.; Lees, C.W.; Franke, A.; D’Amato, M.; Taylor, K.D.; Lee, J.C.; Goyette, P.; Imielinski, M.; Latiano, A.; et al. Meta-analysis identifies 29 additional ulcerative colitis risk loci, increasing the number of confirmed associations to 47. Nat. Genet.2011, 43, 246–252. [Google Scholar] [CrossRef] [PubMed]
- Ellinghaus, D.; Bethune, J.; Petersen, B.S.; Franke, A. The genetics of Crohn’s disease and ulcerative colitis —Status quo and beyond. Scand. J. Gastroenterol.2015, 50, 13–23. [Google Scholar] [CrossRef] [PubMed]
- Jostins, L.; Ripke, S.; Weersma, R.K.; Duerr, R.H.; McGovern, D.P.; Hui, K.Y.; Lee, J.C.; Schumm, L.P.; Sharma, Y.; Anderson, C.A.; et al. Host-microbe interactions have shaped the genetic architecture of inflammatory bowel disease. Nature2012, 491, 119–124. [Google Scholar] [CrossRef] [PubMed]
- Henderson, P.; Russell, R.K.; Satsangi, J.; Wilson, D.C. The changing epidemiology of paediatric inflammatory bowel disease. Aliment. Pharmacol. Ther.2011, 33, 1380–1381. [Google Scholar] [CrossRef] [PubMed]
- Stappenbeck, T.S.; Rioux, J.D.; Mizoguchi, A.; Saitoh, T.; Huett, A.; Darfeuille-Michaud, A.; Wileman, T.; Mizushima, N.; Carding, S.; Akira, S.; et al. Crohn disease: A current perspective on genetics, autophagy and immunity. Autophagy2011, 7, 355–374. [Google Scholar] [CrossRef] [PubMed]
- Wolters, F.L.; Russel, M.G.; Sijbrandij, J.; Schouten, L.J.; Odes, S.; Riis, L.; Munkholm, P.; Langholz, E.; Bodini, P.; O’Morain, C.; et al. Disease outcome of inflammatory bowel disease patients: General outline of a Europe-wide population-based 10-year clinical follow-up study. Scand. J. Gastroenterol.2006. [Google Scholar] [CrossRef] [PubMed]
- Amre, D.K.; D’Souza, S.; Morgan, K.; Seidman, G.; Lambrette, P.; Grimard, G.; Israel, D.; Mack, D.; Ghadirian, P.; Deslandres, C.; et al. Imbalances in dietary consumption of fatty acids, vegetables, and fruits are associated with risk for Crohn’s disease in children. Am. J. Gastroenterol.2007, 102, 2016–2025. [Google Scholar] [CrossRef] [PubMed]
- Kaufmann, H.J.; Taubin, H.L. Nonsteroidal anti-inflammatory drugs activate quiescent inflammatory bowel disease. Ann. Intern. Med.1987, 107, 513–516. [Google Scholar] [CrossRef] [PubMed]
- Helzer, J.E. The impact of combat on later alcohol use by Vietnam veterans. J. Psychoactive Drugs1984, 16, 183–191. [Google Scholar] [CrossRef] [PubMed]
- Knights, D.; Lassen, K.G.; Xavier, R.J. Advances in inflammatory bowel disease pathogenesis: Linking host genetics and the microbiome. Gut2013, 62, 1505–1510. [Google Scholar] [CrossRef] [PubMed]
- Lee, K.N.; Lee, O.Y. Intestinal microbiota in pathophysiology and management of irritable bowel syndrome. World J. Gastroenterol.2014, 20, 8886–8897. [Google Scholar] [CrossRef] [PubMed]
- Whitehead, W.E.; Palsson, O.; Jones, K.R. Systematic review of the comorbidity of irritable bowel syndrome with other disorders: What are the causes and implications? Gastroenterology2002, 122, 1140–1156. [Google Scholar] [CrossRef] [PubMed]
- Savage, D.C. Microbial ecology of the gastrointestinal tract. Annu. Rev. Microbiol.1977, 31, 107–133. [Google Scholar] [CrossRef] [PubMed]
- Sender, R.; Fuchs, S.; Milo, R. Revised estimates for the number of human and bacteria cells in the body. bioRxiv2016. [Google Scholar] [CrossRef]
- Berg, R.D. The indigenous gastrointestinal microflora. Trends Microbiol.1996, 4, 430–435. [Google Scholar] [CrossRef]
- Ley, R.E.; Peterson, D.A.; Gordon, J.I. Ecological and evolutionary forces shaping microbial diversity in the human intestine. Cell2006, 124, 837–848. [Google Scholar] [CrossRef] [PubMed]
- Qin, J.; Li, R.; Raes, J.; Arumugam, M.; Burgdorf, K.S.; Manichanh, C.; Nielsen, T.; Pons, N.; Levenez, F.; Yamada, T.; et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature2010, 464, 59–65. [Google Scholar] [CrossRef] [PubMed]
- Eckburg, P.B.; Relman, D.A. The role of microbes in Crohn’s disease. Clin. Infect. Dis.2007, 44, 256–262. [Google Scholar] [CrossRef] [PubMed]
- Karlsson, F.H.; Fak, F.; Nookaew, I.; Tremaroli, V.; Fagerberg, B.; Petranovic, D.; Backhed, F.; Nielsen, J. Symptomatic atherosclerosis is associated with an altered gut metagenome. Nat. Commun.2012. [Google Scholar] [CrossRef] [PubMed]
- Arumugam, M.; Raes, J.; Pelletier, E.; Le Paslier, D.; Yamada, T.; Mende, D.R.; Fernandes, G.R.; Tap, J.; Bruls, T.; Batto, J.M.; et al. Enterotypes of the human gut microbiome. Nature2011, 473, 174–180. [Google Scholar] [CrossRef] [PubMed]
- Relman, D.A. The human microbiome: Ecosystem resilience and health. Nutr. Rev.2012, 70, S2–S9. [Google Scholar] [CrossRef] [PubMed]
- Flint, H.J. Microbiology: Antibiotics and adiposity. Nature2012, 488, 601–602. [Google Scholar] [CrossRef] [PubMed]
- Walker, A.W.; Sanderson, J.D.; Churcher, C.; Parkes, G.C.; Hudspith, B.N.; Rayment, N.; Brostoff, J.; Parkhill, J.; Dougan, G.; Petrovska, L. High-throughput clone library analysis of the mucosa-associated microbiota reveals dysbiosis and differences between inflamed and non-inflamed regions of the intestine in inflammatory bowel disease. BMC Microbiol.2011. [Google Scholar] [CrossRef] [PubMed]
- Duncan, S.H.; Belenguer, A.; Holtrop, G.; Johnstone, A.M.; Flint, H.J.; Lobley, G.E. Reduced dietary intake of carbohydrates by obese subjects results in decreased concentrations of butyrate and butyrate-producing bacteria in feces. Appl. Environ. Microbiol.2007, 73, 1073–1078. [Google Scholar] [CrossRef] [PubMed]
- Russell, W.R.; Gratz, S.W.; Duncan, S.H.; Holtrop, G.; Ince, J.; Scobbie, L.; Duncan, G.; Johnstone, A.M.; Lobley, G.E.; Wallace, R.J.; et al. High-protein, reduced-carbohydrate weight-loss diets promote metabolite profiles likely to be detrimental to colonic health. Am. J. Clin. Nutr.2011, 93, 1062–1072. [Google Scholar] [CrossRef] [PubMed]
- Greiner, A.K.; Papineni, R.V.; Umar, S. Chemoprevention in gastrointestinal physiology and disease. Natural products and microbiome. Am. J. Physiol. Gastrointest. Liver Physiol.2014, 307, G1–G15. [Google Scholar] [CrossRef] [PubMed]
- Zaneveld, J.; Turnbaugh, P.J.; Lozupone, C.; Ley, R.E.; Hamady, M.; Gordon, J.I.; Knight, R. Host-bacterial coevolution and the search for new drug targets. Curr. Opin. Chem. Biol.2008, 12, 109–114. [Google Scholar] [CrossRef] [PubMed]
- Hamer, H.M.; Jonkers, D.M.; Bast, A.; Vanhoutvin, S.A.; Fischer, M.A.; Kodde, A.; Troost, F.J.; Venema, K.; Brummer, R.J. Butyrate modulates oxidative stress in the colonic mucosa of healthy humans. Clin. Nutr.2009, 28, 88–93. [Google Scholar] [CrossRef] [PubMed]
- Hooper, L.V. Bacterial contributions to mammalian gut development. Trends Microbiol.2004, 12, 129–134. [Google Scholar] [CrossRef] [PubMed]
- Hooper, L.V.; Gordon, J.I. Commensal host-bacterial relationships in the gut. Science2001, 292, 1115–1118. [Google Scholar] [CrossRef] [PubMed]
- Frick, J.S.; Autenrieth, I.B. The gut microflora and its variety of roles in health and disease. Curr. Top. Microbiol. Immunol.2013, 358, 273–289. [Google Scholar] [PubMed]
- Costello, E.K.; Stagaman, K.; Dethlefsen, L.; Bohannan, B.J.; Relman, D.A. The application of ecological theory toward an understanding of the human microbiome. Science2012, 336, 1255–1262. [Google Scholar] [CrossRef] [PubMed]
- Yatsunenko, T.; Rey, F.E.; Manary, M.J.; Trehan, I.; Dominguez-Bello, M.G.; Contreras, M.; Magris, M.; Hidalgo, G.; Baldassano, R.N.; Anokhin, A.P.; et al. Human gut microbiome viewed across age and geography. Nature2012, 486, 222–227. [Google Scholar] [CrossRef] [PubMed]
- Gosalbes, M.J.; Abellan, J.J.; Durban, A.; Perez-Cobas, A.E.; Latorre, A.; Moya, A. Metagenomics of human microbiome: Beyond 16s rDNA. Clin. Microbiol. Infect.2012, 18, 47–49. [Google Scholar] [CrossRef] [PubMed]
- Holmes, E.; Li, J.V.; Marchesi, J.R.; Nicholson, J.K. Gut microbiota composition and activity in relation to host metabolic phenotype and disease risk. Cell Metab.2012, 16, 559–564. [Google Scholar] [CrossRef] [PubMed]
- Comito, D.; Cascio, A.; Romano, C. Microbiota biodiversity in inflammatory bowel disease. Ital. J. Pediatr.2014. [Google Scholar] [CrossRef] [PubMed]
- Sartor, R.B. Targeting enteric bacteria in treatment of inflammatory bowel diseases: Why, how, and when. Curr. Opin. Gastroenterol.2003, 19, 358–365. [Google Scholar] [CrossRef] [PubMed]
- Taurog, J.D.; Richardson, J.A.; Croft, J.T.; Simmons, W.A.; Zhou, M.; Fernandez-Sueiro, J.L.; Balish, E.; Hammer, R.E. The germfree state prevents development of gut and joint inflammatory disease in hla-b27 transgenic rats. J. Exp. Med.1994, 180, 2359–2364. [Google Scholar] [CrossRef] [PubMed]
- Danese, S.; Sans, M.; Fiocchi, C. Inflammatory bowel disease: The role of environmental factors. Autoimmun. Rev.2004, 3, 394–400. [Google Scholar] [CrossRef] [PubMed]
- Kamada, N.; Kao, J.Y. The tuning of the gut nervous system by commensal microbiota. Gastroenterology2013, 145, 1193–1196. [Google Scholar] [CrossRef] [PubMed]
- Corridoni, D.; Pastorelli, L.; Mattioli, B.; Locovei, S.; Ishikawa, D.; Arseneau, K.O.; Chieppa, M.; Cominelli, F.; Pizarro, T.T. Probiotic bacteria regulate intestinal epithelial permeability in experimental ileitis by a TNF-dependent mechanism. PLoS ONE2012, 7, e42067. [Google Scholar] [CrossRef] [PubMed]
- Gionchetti, P.; Rizzello, F.; Habal, F.; Morselli, C.; Amadini, C.; Romagnoli, R.; Campieri, M. Standard treatment of ulcerative colitis. Dig. Dis.2003, 21, 157–167. [Google Scholar] [CrossRef] [PubMed]
- Hansen, R.; Thomson, J.M.; El-Omar, E.M.; Hold, G.L. The role of infection in the aetiology of inflammatory bowel disease. J. Gastroenterol.2010, 45, 266–276. [Google Scholar] [CrossRef] [PubMed]
- Frank, D.N.; St Amand, A.L.; Feldman, R.A.; Boedeker, E.C.; Harpaz, N.; Pace, N.R. Molecular-phylogenetic characterization of microbial community imbalances in human inflammatory bowel diseases. Proc. Natl. Acad. Sci. USA2007, 104, 13780–13785. [Google Scholar] [CrossRef] [PubMed]
- Sokol, H.; Lepage, P.; Seksik, P.; Dore, J.; Marteau, P. Temperature gradient gel electrophoresis of fecal 16s rRNA reveals active Escherichia coli in the microbiota of patients with ulcerative colitis. J. Clin. Microbiol.2006, 44, 3172–3177. [Google Scholar] [CrossRef] [PubMed]
- Peterson, D.A.; Frank, D.N.; Pace, N.R.; Gordon, J.I. Metagenomic approaches for defining the pathogenesis of inflammatory bowel diseases. Cell Host Microbe2008, 3, 417–427. [Google Scholar] [CrossRef] [PubMed]
- Mondot, S.; Kang, S.; Furet, J.P.; Aguirre de Carcer, D.; McSweeney, C.; Morrison, M.; Marteau, P.; Dore, J.; Leclerc, M. Highlighting new phylogenetic specificities of Crohn’s disease microbiota. Inflamm. Bowel Dis.2011, 17, 185–192. [Google Scholar] [CrossRef] [PubMed]
- Joossens, M.; Huys, G.; Cnockaert, M.; de Preter, V.; Verbeke, K.; Rutgeerts, P.; Vandamme, P.; Vermeire, S. Dysbiosis of the faecal microbiota in patients with Crohn’s disease and their unaffected relatives. Gut2011, 60, 631–637. [Google Scholar] [CrossRef] [PubMed]
- Prindiville, T.P.; Sheikh, R.A.; Cohen, S.H.; Tang, Y.J.; Cantrell, M.C.; Silva, J., Jr. Bacteroides fragilis enterotoxin gene sequences in patients with inflammatory bowel disease. Emerg. Infect. Dis.2000, 6, 171–174. [Google Scholar] [CrossRef] [PubMed]
- Neut, C.; Bulois, P.; Desreumaux, P.; Membre, J.M.; Lederman, E.; Gambiez, L.; Cortot, A.; Quandalle, P.; van Kruiningen, H.; Colombel, J.F. Changes in the bacterial flora of the neoterminal ileum after ileocolonic resection for Crohn’s disease. Am. J. Gastroenterol.2002, 97, 939–946. [Google Scholar] [CrossRef] [PubMed]
- Andoh, A.; Kuzuoka, H.; Tsujikawa, T.; Nakamura, S.; Hirai, F.; Suzuki, Y.; Matsui, T.; Fujiyama, Y.; Matsumoto, T. Multicenter analysis of fecal microbiota profiles in Japanese patients with Crohn’s disease. J. Gastroenterol.2012, 47, 1298–1307. [Google Scholar] [CrossRef] [PubMed]
- Seksik, P.; Rigottier-Gois, L.; Gramet, G.; Sutren, M.; Pochart, P.; Marteau, P.; Jian, R.; Dore, J. Alterations of the dominant faecal bacterial groups in patients with Crohn’s disease of the colon. Gut2003, 52, 237–242. [Google Scholar] [CrossRef] [PubMed]
- Lucke, K.; Miehlke, S.; Jacobs, E.; Schuppler, M. Prevalence of bacteroides and Prevotella spp. In ulcerative colitis. J. Med. Microbiol.2006, 55, 617–624. [Google Scholar] [CrossRef] [PubMed]
- Mukhopadhya, I.; Hansen, R.; El-Omar, E.M.; Hold, G.L. IBD-what role do proteobacteria play? Nat. Rev. Gastroenterol. Hepatol.2012, 9, 219–230. [Google Scholar] [CrossRef] [PubMed]
- Rehman, A.; Lepage, P.; Nolte, A.; Hellmig, S.; Schreiber, S.; Ott, S.J. Transcriptional activity of the dominant gut mucosal microbiota in chronic inflammatory bowel disease patients. J. Med. Microbiol.2010, 59, 1114–1122. [Google Scholar] [CrossRef] [PubMed]
- Baumgart, M.; Dogan, B.; Rishniw, M.; Weitzman, G.; Bosworth, B.; Yantiss, R.; Orsi, R.H.; Wiedmann, M.; McDonough, P.; Kim, S.G.; et al. Culture independent analysis of ileal mucosa reveals a selective increase in invasive escherichia coli of novel phylogeny relative to depletion of clostridiales in crohn’s disease involving the ileum. ISME J.2007, 1, 403–418. [Google Scholar] [CrossRef] [PubMed]
- Gophna, U.; Sommerfeld, K.; Gophna, S.; Doolittle, W.F.; Veldhuyzen van Zanten, S.J. Differences between tissue-associated intestinal microfloras of patients with Crohn’s disease and ulcerative colitis. J. Clin. Microbiol.2006, 44, 4136–4141. [Google Scholar] [CrossRef] [PubMed]
- Lupp, C.; Robertson, M.L.; Wickham, M.E.; Sekirov, I.; Champion, O.L.; Gaynor, E.C.; Finlay, B.B. Host-mediated inflammation disrupts the intestinal microbiota and promotes the overgrowth of enterobacteriaceae. Cell Host Microbe2007, 2, 119–129. [Google Scholar] [CrossRef] [PubMed]
- Willing, B.; Halfvarson, J.; Dicksved, J.; Rosenquist, M.; Jarnerot, G.; Engstrand, L.; Tysk, C.; Jansson, J.K. Twin studies reveal specific imbalances in the mucosa-associated microbiota of patients with ileal Crohn’s disease. Inflamm. Bowel Dis.2009, 15, 653–660. [Google Scholar] [CrossRef] [PubMed]
- Darfeuille-Michaud, A.; Neut, C.; Barnich, N.; Lederman, E.; di Martino, P.; Desreumaux, P.; Gambiez, L.; Joly, B.; Cortot, A.; Colombel, J.F. Presence of adherent Escherichia coli strains in ileal mucosa of patients with Crohn’s disease. Gastroenterology1998, 115, 1405–1413. [Google Scholar] [CrossRef]
- Boudeau, J.; Glasser, A.L.; Masseret, E.; Joly, B.; Darfeuille-Michaud, A. Invasive ability of an Escherichia coli strain isolated from the ileal mucosa of a patient with Crohn’s disease. Infect. Immun.1999, 67, 4499–4509. [Google Scholar] [PubMed]
- Darfeuille-Michaud, A.; Boudeau, J.; Bulois, P.; Neut, C.; Glasser, A.L.; Barnich, N.; Bringer, M.A.; Swidsinski, A.; Beaugerie, L.; Colombel, J.F. High prevalence of adherent-invasive Escherichia coli associated with ileal mucosa in Crohn’s disease. Gastroenterology2004, 127, 412–421. [Google Scholar] [CrossRef] [PubMed]
- Naser, S.A.; Sagramsingh, S.R.; Naser, A.S.; Thanigachalam, S. Mycobacterium avium subspecies paratuberculosis causes Crohn’s disease in some inflammatory bowel disease patients. World J. Gastroenterol.2014, 20, 7403–7415. [Google Scholar] [CrossRef] [PubMed]
- Nazareth, N.; Magro, F.; Machado, E.; Ribeiro, T.G.; Martinho, A.; Rodrigues, P.; Alves, R.; Macedo, G.N.; Gracio, D.; Coelho, R.; et al. Prevalence of Mycobacterium avium subsp. paratuberculosis and Escherichia coli in blood samples from patients with inflammatory bowel disease. Med. Microbiol. Immunol.2015, 204, 681–692. [Google Scholar] [CrossRef] [PubMed]
- Mahendran, V.; Riordan, S.M.; Grimm, M.C.; Tran, T.A.; Major, J.; Kaakoush, N.O.; Mitchell, H.; Zhang, L. Prevalence of campylobacter species in adult Crohn’s disease and the preferential colonization sites of campylobacter species in the human intestine. PLoS ONE2011, 6, e25417. [Google Scholar] [CrossRef] [PubMed]
- Man, S.M.; Zhang, L.; Day, A.S.; Leach, S.T.; Lemberg, D.A.; Mitchell, H. Campylobacter concisus and other campylobacter species in children with newly diagnosed Crohn’s disease. Inflamm. Bowel Dis.2010, 16, 1008–1016. [Google Scholar] [CrossRef] [PubMed]
- Zhang, L.; Man, S.M.; Day, A.S.; Leach, S.T.; Lemberg, D.A.; Dutt, S.; Stormon, M.; Otley, A.; O’Loughlin, E.V.; Magoffin, A.; et al. Detection and isolation of campylobacter species other than C. jejuni from children with Crohn’s disease. J. Clin. Microbiol.2009, 47, 453–455. [Google Scholar] [CrossRef] [PubMed]
- Kim, H.; Rhee, S.H.; Pothoulakis, C.; Lamont, J.T. Inflammation and apoptosis in clostridium difficile enteritis is mediated by PGE2 up-regulation of Fas ligand. Gastroenterology2007, 133, 875–886. [Google Scholar] [CrossRef] [PubMed]
- Issa, M.; Vijayapal, A.; Graham, M.B.; Beaulieu, D.B.; Otterson, M.F.; Lundeen, S.; Skaros, S.; Weber, L.R.; Komorowski, R.A.; Knox, J.F.; et al. Impact of clostridium difficile on inflammatory bowel disease. Clin. Gastroenterol.Hepatol.2007, 5, 345–351. [Google Scholar] [CrossRef] [PubMed]
- Rabizadeh, S.; Rhee, K.J.; Wu, S.; Huso, D.; Gan, C.M.; Golub, J.E.; Wu, X.; Zhang, M.; Sears, C.L. Enterotoxigenic Bacteroides fragilis: A potential instigator of colitis. Inflamm. Bowel Dis.2007, 13, 1475–1483. [Google Scholar] [CrossRef] [PubMed]
- Dominguez-Bello, M.G.; Blaser, M.J.; Ley, R.E.; Knight, R. Development of the human gastrointestinal microbiota and insights from high-throughput sequencing. Gastroenterology2011, 140, 1713–1719. [Google Scholar] [CrossRef] [PubMed]
- Palmer, C.; Bik, E.M.; DiGiulio, D.B.; Relman, D.A.; Brown, P.O. Development of the human infant intestinal microbiota. PLoS Biol.2007, 5, e177. [Google Scholar] [CrossRef] [PubMed]
- Koenig, J.E.; Spor, A.; Scalfone, N.; Fricker, A.D.; Stombaugh, J.; Knight, R.; Angenent, L.T.; Ley, R.E. Succession of microbial consortia in the developing infant gut microbiome. Proc. Natl. Acad. Sci. USA2011, 108, 4578–4585. [Google Scholar] [CrossRef] [PubMed]
- Schwartz, S.; Friedberg, I.; Ivanov, I.V.; Davidson, L.A.; Goldsby, J.S.; Dahl, D.B.; Herman, D.; Wang, M.; Donovan, S.M.; Chapkin, R.S. A metagenomic study of diet-dependent interaction between gut microbiota and host in infants reveals differences in immune response. Genome Biol.2012. [Google Scholar] [CrossRef] [PubMed]
- Roger, L.C.; Costabile, A.; Holland, D.T.; Hoyles, L.; McCartney, A.L. Examination of faecal bifidobacterium populations in breast- and formula-fed infants during the first 18 months of life. Microbiology2010, 156, 3329–3341. [Google Scholar] [CrossRef] [PubMed]
- Fallani, M.; Amarri, S.; Uusijarvi, A.; Adam, R.; Khanna, S.; Aguilera, M.; Gil, A.; Vieites, J.M.; Norin, E.; Young, D.; et al. Determinants of the human infant intestinal microbiota after the introduction of first complementary foods in infant samples from five European centres. Microbiology2011, 157, 1385–1392. [Google Scholar] [CrossRef] [PubMed]
- Fallani, M.; Young, D.; Scott, J.; Norin, E.; Amarri, S.; Adam, R.; Aguilera, M.; Khanna, S.; Gil, A.; Edwards, C.A.; et al. Intestinal microbiota of 6-week-old infants across Europe: Geographic influence beyond delivery mode, breast-feeding, and antibiotics. J. Pediatr. Gastroenterol. Nutr.2010, 51, 77–84. [Google Scholar] [CrossRef] [PubMed]
- Chapman-Kiddell, C.A.; Davies, P.S.; Gillen, L.; Radford-Smith, G.L. Role of diet in the development of inflammatory bowel disease. Inflamm. Bowel Dis.2010, 16, 137–151. [Google Scholar] [CrossRef] [PubMed]
- Hou, J.K.; Abraham, B.; El-Serag, H. Dietary intake and risk of developing inflammatory bowel disease: A systematic review of the literature. Am. J. Gastroenterol.2011, 106, 563–573. [Google Scholar] [CrossRef] [PubMed]
- D’Haens, G.R.; Sartor, R.B.; Silverberg, M.S.; Petersson, J.; Rutgeerts, P. Future directions in inflammatory bowel disease management. J. Crohns Colitis2014, 8, 726–734. [Google Scholar] [CrossRef] [PubMed]
- Richman, E.; Rhodes, J.M. Review article: Evidence-based dietary advice for patients with inflammatory bowel disease. Aliment. Pharmacol. Ther.2013, 38, 1156–1171. [Google Scholar] [CrossRef] [PubMed]
- De Filippo, C.; Cavalieri, D.; di Paola, M.; Ramazzotti, M.; Poullet, J.B.; Massart, S.; Collini, S.; Pieraccini, G.; Lionetti, P. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa. Proc. Natl. Acad. Sci. USA2010, 107, 14691–14696. [Google Scholar] [CrossRef] [PubMed]
- Spooren, C.E.; Pierik, M.J.; Zeegers, M.P.; Feskens, E.J.; Masclee, A.A.; Jonkers, D.M. Review article: The association of diet with onset and relapse in patients with inflammatory bowel disease. Aliment. Pharmacol. Ther.2013, 38, 1172–1187. [Google Scholar] [CrossRef] [PubMed]
- Hou, J.K.; Lee, D.; Lewis, J. Diet and inflammatory bowel disease: Review of patient-targeted recommendations. Clin. Gastroenterol. Hepatol.2014, 12, 1592–1600. [Google Scholar] [CrossRef] [PubMed]
- Ananthakrishnan, A.N. Environmental risk factors for inflammatory bowel disease. Gastroenterol. Hepatol.2013, 9, 367–374. [Google Scholar] [CrossRef] [PubMed]
- Durchschein, F.; Petritsch, W.; Hammer, H.F. Diet therapy for inflammatory bowel diseases: The established and the new. World J. Gastroenterol.2016, 22, 2179–2194. [Google Scholar] [PubMed]
- Walton, C.; Montoya, M.P.; Fowler, D.P.; Turner, C.; Jia, W.; Whitehead, R.N.; Griffiths, L.; Waring, R.H.; Ramsden, D.B.; Cole, J.A.; et al. Enteral feeding reduces metabolic activity of the intestinal microbiome in Crohn’s disease: An observational study. Eur. J. Clin. Nutr.2016. [Google Scholar] [CrossRef] [PubMed]
- Kaakoush, N.O.; Day, A.S.; Leach, S.T.; Lemberg, D.A.; Nielsen, S.; Mitchell, H.M. Effect of exclusive enteral nutrition on the microbiota of children with newly diagnosed Crohn’s disease. Clin. Transl. Gastroenterol.2015. [Google Scholar] [CrossRef] [PubMed]
- Day, A.S.; Lopez, R.N. Exclusive enteral nutrition in children with Crohn’s disease. World J. Gastroenterol.2015, 21, 6809–6816. [Google Scholar] [PubMed]
- Heuschkel, R.B.; Menache, C.C.; Megerian, J.T.; Baird, A.E. Enteral nutrition and corticosteroids in the treatment of acute Crohn’s disease in children. J. Pediatr. Gastroenterol. Nutr.2000, 31, 8–15. [Google Scholar] [CrossRef] [PubMed]
- Afzal, N.A.; Davies, S.; Paintin, M.; Arnaud-Battandier, F.; Walker-Smith, J.A.; Murch, S.; Heuschkel, R.; Fell, J. Colonic Crohn’s disease in children does not respond well to treatment with enteral nutrition if the ileum is not involved. Dig. Dis. Sci.2005, 50, 1471–1475. [Google Scholar] [CrossRef] [PubMed]
- Cui, B.; Li, P.; Xu, L.; Peng, Z.; Xiang, J.; He, Z.; Zhang, T.; Ji, G.; Nie, Y.; Wu, K.; et al. Step-up fecal microbiota transplantation (FMT) strategy. Gut Microbes2016. [Google Scholar] [CrossRef] [PubMed]
- Harris, J.K.; El Kasmi, K.C.; Anderson, A.L.; Devereaux, M.W.; Fillon, S.A.; Robertson, C.E.; Wagner, B.D.; Stevens, M.J.; Pace, N.R.; Sokol, R.J. Specific microbiome changes in a mouse model of parenteral nutrition associated liver injury and intestinal inflammation. PLoS ONE2014, 9, e110396. [Google Scholar] [CrossRef] [PubMed]
- Devkota, S.; Chang, E.B. Diet-induced expansion of pathobionts in experimental colitis: Implications for tailored therapies. Gut Microbes2013, 4, 172–174. [Google Scholar] [CrossRef] [PubMed]
- Devkota, S.; Wang, Y.; Musch, M.W.; Leone, V.; Fehlner-Peach, H.; Nadimpalli, A.; Antonopoulos, D.A.; Jabri, B.; Chang, E.B. Dietary-fat-induced taurocholic acid promotes pathobiont expansion and colitis in Il10 −/− mice. Nature2012, 487, 104–108. [Google Scholar] [CrossRef] [PubMed]
- Fiehn, O. Combining genomics, metabolome analysis, and biochemical modelling to understand metabolic networks. Comp. Funct Genomics2001, 2, 155–168. [Google Scholar] [CrossRef] [PubMed]
- Griffin, J.L.; Nicholls, A.W. Metabolomics as a functional genomic tool for understanding lipid dysfunction in diabetes, obesity and related disorders. Pharmacogenomics2006, 7, 1095–1107. [Google Scholar] [CrossRef] [PubMed]
- Nicholson, J.K.; Lindon, J.C.; Holmes, E. ”Metabonomics”: Understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica1999, 29, 1181–1189. [Google Scholar] [CrossRef] [PubMed]
- Maurice, C.F.; Haiser, H.J.; Turnbaugh, P.J. Xenobiotics shape the physiology and gene expression of the active human gut microbiome. Cell2013, 152, 39–50. [Google Scholar] [CrossRef] [PubMed]
- McNulty, N.P.; Yatsunenko, T.; Hsiao, A.; Faith, J.J.; Muegge, B.D.; Goodman, A.L.; Henrissat, B.; Oozeer, R.; Cools-Portier, S.; Gobert, G.; et al. The impact of a consortium of fermented milk strains on the gut microbiome of gnotobiotic mice and monozygotic twins. Sci. Transl. Med.2011. [Google Scholar] [CrossRef] [PubMed]
- Marcobal, A.; Kashyap, P.C.; Nelson, T.A.; Aronov, P.A.; Donia, M.S.; Spormann, A.; Fischbach, M.A.; Sonnenburg, J.L. A metabolomic view of how the human gut microbiota impacts the host metabolome using humanized and gnotobiotic mice. Isme J.2013, 7, 1933–1943. [Google Scholar] [CrossRef] [PubMed]
- Fiehn, O. Metabolomics—The link between genotypes and phenotypes. Plant Mol. Biol.2002, 48, 155–171. [Google Scholar] [CrossRef] [PubMed]
- Kim, J.K.; Bamba, T.; Harada, K.; Fukusaki, E.; Kobayashi, A. Time-course metabolic profiling in Arabidopsis thaliana cell cultures after salt stress treatment. J. Exp. Bot.2007, 58, 415–424. [Google Scholar] [CrossRef] [PubMed]
- Borner, J.; Buchinger, S.; Schomburg, D. A high-throughput method for microbial metabolome analysis using gas chromatography/mass spectrometry. Anal. Biochem.2007, 367, 143–151. [Google Scholar] [CrossRef] [PubMed]
- Kind, T.; Tolstikov, V.; Fiehn, O.; Weiss, R.H. A comprehensive urinary metabolomic approach for identifying kidney cancerr. Anal. Biochem.2007, 363, 185–195. [Google Scholar] [CrossRef] [PubMed]
- Parveen, I.; Moorby, J.M.; Fraser, M.D.; Allison, G.G.; Kopka, J. Application of gas chromatography-mass spectrometry metabolite profiling techniques to the analysis of heathland plant diets of sheep. J. Agric. Food Chem.2007, 55, 1129–1138. [Google Scholar] [CrossRef] [PubMed]
- Haiser, H.J.; Turnbaugh, P.J. Is it time for a metagenomic basis of therapeutics? Science2012, 336, 1253–1255. [Google Scholar] [CrossRef] [PubMed]
- Shiomi, Y.; Nishiumi, S.; Ooi, M.; Hatano, N.; Shinohara, M.; Yoshie, T.; Kondo, Y.; Furumatsu, K.; Shiomi, H.; Kutsumi, H.; et al. Gcms-based metabolomic study in mice with colitis induced by dextran sulfate sodium. Inflamm. Bowel Dis.2011, 17, 2261–2274. [Google Scholar] [CrossRef] [PubMed]
- Dawiskiba, T.; Deja, S.; Mulak, A.; Zabek, A.; Jawien, E.; Pawelka, D.; Banasik, M.; Mastalerz-Migas, A.; Balcerzak, W.; Kaliszewski, K.; et al. Serum and urine metabolomic fingerprinting in diagnostics of inflammatory bowel diseases. World J. Gastroenterol.2014, 20, 163–174. [Google Scholar] [CrossRef] [PubMed]
- De Preter, V.; Machiels, K.; Joossens, M.; Arijs, I.; Matthys, C.; Vermeire, S.; Rutgeerts, P.; Verbeke, K. Faecal metabolite profiling identifies medium-chain fatty acids as discriminating compounds in IBD. Gut2015, 64, 447–458. [Google Scholar] [CrossRef] [PubMed]
- McIntosh, K.; Reed, D.E.; Schneider, T.; Dang, F.; Keshteli, A.H.; de Palma, G.; Madsen, K.; Bercik, P.; Vanner, S. Fodmaps alter symptoms and the metabolome of patients with IBS: A randomised controlled trial. Gut2016. [Google Scholar] [CrossRef] [PubMed]
- Machiels, K.; Joossens, M.; Sabino, J.; de Preter, V.; Arijs, I.; Eeckhaut, V.; Ballet, V.; Claes, K.; van Immerseel, F.; Verbeke, K.; et al. A decrease of the butyrate-producing species Roseburia hominis and Faecalibacterium prausnitzii defines dysbiosis in patients with ulcerative colitis. Gut2014, 63, 1275–1283. [Google Scholar] [CrossRef] [PubMed]
- Wang, W.; Chen, L.; Zhou, R.; Wang, X.; Song, L.; Huang, S.; Wang, G.; Xia, B. Increased proportions of bifidobacterium and the lactobacillus group and loss of butyrate-producing bacteria in inflammatory bowel disease. J. Clin. Microbiol.2014, 52, 398–406. [Google Scholar] [CrossRef] [PubMed]
- Garber, K. Drugging the gut microbiome. Nat. Biotechnol.2015, 33, 228–231. [Google Scholar] [CrossRef] [PubMed]
- Spiller, R. Irritable bowel syndrome: New insights into symptom mechanisms and advances in treatment. F1000Research2016. [Google Scholar] [CrossRef] [PubMed]
- Balasubramanian, K.; Kumar, S.; Singh, R.R.; Sharma, U.; Ahuja, V.; Makharia, G.K.; Jagannathan, N.R. Metabolism of the colonic mucosa in patients with inflammatory bowel diseases: An in vitro proton magnetic resonance spectroscopy study. Magn. Reson. Imaging2009, 27, 79–86. [Google Scholar] [CrossRef] [PubMed]
- Bezabeh, T.; Somorjai, R.L.; Smith, I.C.; Nikulin, A.E.; Dolenko, B.; Bernstein, C.N. The use of 1 H magnetic resonance spectroscopy in inflammatory bowel diseases: Distinguishing ulcerative colitis from Crohn’s disease. Am. J. Gastroenterol.2001, 96, 442–448. [Google Scholar] [CrossRef] [PubMed]
- Bach, S.P.; Mortensen, N.J. Ileal pouch surgery for ulcerative colitis. World J. Gastroenterol.2007, 13, 3288–3300. [Google Scholar] [CrossRef] [PubMed]
- Odze, R.D. A contemporary and critical appraisal of ”indeterminate colitis”. Mod. Pathol.2015, 28, S30–S46. [Google Scholar] [CrossRef] [PubMed]
- Tontini, G.E.; Vecchi, M.; Pastorelli, L.; Neurath, M.F.; Neumann, H. Differential diagnosis in inflammatory bowel disease colitis: State of the art and future perspectives. World J. Gastroenterol.2015, 21, 21–46. [Google Scholar] [CrossRef] [PubMed]
- Dotan, I. New serologic markers for inflammatory bowel disease diagnosis. Dig. Dis.2010, 28, 418–423. [Google Scholar] [CrossRef] [PubMed]
- Van Schaik, F.D.; Oldenburg, B.; Hart, A.R.; Siersema, P.D.; Lindgren, S.; Grip, O.; Teucher, B.; Kaaks, R.; Bergmann, M.M.; Boeing, H.; et al. Serological markers predict inflammatory bowel disease years before the diagnosis. Gut2013, 62, 683–688. [Google Scholar] [CrossRef] [PubMed]
- Zholudev, A.; Zurakowski, D.; Young, W.; Leichtner, A.; Bousvaros, A. Serologic testing with ANCA, ASCA, and anti-OmpC in children and young adults with Crohn’s disease and ulcerative colitis: Diagnostic value and correlation with disease phenotype. Am. J. Gastroenterol.2004, 99, 2235–2241. [Google Scholar] [CrossRef] [PubMed]
- Kuna, A.T. Serological markers of inflammatory bowel disease. Biochem. Med.2013, 23, 28–42. [Google Scholar] [CrossRef]
- Schicho, R.; Shaykhutdinov, R.; Ngo, J.; Nazyrova, A.; Schneider, C.; Panaccione, R.; Kaplan, G.G.; Vogel, H.J.; Storr, M. Quantitative metabolomic profiling of serum, plasma, and urine by 1 H NMR spectroscopy discriminates between patients with inflammatory bowel disease and healthy individuals. J. Proteome Res.2012, 11, 3344–3357. [Google Scholar] [CrossRef] [PubMed]
- Williams, H.R.; Willsmore, J.D.; Cox, I.J.; Walker, D.G.; Cobbold, J.F.; Taylor-Robinson, S.D.; Orchard, T.R. Serum metabolic profiling in inflammatory bowel disease. Dig. Dis. Sci.2012, 57, 2157–2165. [Google Scholar] [CrossRef] [PubMed]
- Stephens, N.S.; Siffledeen, J.; Su, X.; Murdoch, T.B.; Fedorak, R.N.; Slupsky, C.M. Urinary NMR metabolomic profiles discriminate inflammatory bowel disease from healthy. J. Crohns Colitis2013, 7, e42–e48. [Google Scholar] [CrossRef] [PubMed]
- Le Gall, G.; Noor, S.O.; Ridgway, K.; Scovell, L.; Jamieson, C.; Johnson, I.T.; Colquhoun, I.J.; Kemsley, E.K.; Narbad, A. Metabolomics of fecal extracts detects altered metabolic activity of gut microbiota in ulcerative colitis and irritable bowel syndrome. J. Proteome Res.2011, 10, 4208–4218. [Google Scholar] [CrossRef] [PubMed]
- Bjerrum, J.T.; Nielsen, O.H.; Hao, F.; Tang, H.; Nicholson, J.K.; Wang, Y.; Olsen, J. Metabonomics in ulcerative colitis: Diagnostics, biomarker identification, and insight into the pathophysiology. J. Proteome Res.2010, 9, 954–962. [Google Scholar] [CrossRef] [PubMed]
- Williams, H.R.; Cox, I.J.; Walker, D.G.; North, B.V.; Patel, V.M.; Marshall, S.E.; Jewell, D.P.; Ghosh, S.; Thomas, H.J.; Teare, J.P.; et al. Characterization of inflammatory bowel disease with urinary metabolic profiling. Am. J. Gastroenterol.2009, 104, 1435–1444. [Google Scholar] [CrossRef] [PubMed]
- Jansson, J.; Willing, B.; Lucio, M.; Fekete, A.; Dicksved, J.; Halfvarson, J.; Tysk, C.; Schmitt-Kopplin, P. Metabolomics reveals metabolic biomarkers of Crohn’s disease. PLoS ONE2009, 4, e6386. [Google Scholar] [CrossRef] [PubMed]
- Sokol, H.; Seksik, P.; Rigottier-Gois, L.; Lay, C.; Lepage, P.; Podglajen, I.; Marteau, P.; Dore, J. Specificities of the fecal microbiota in inflammatory bowel disease. Inflamm. Bowel Dis.2006, 12, 106–111. [Google Scholar] [CrossRef] [PubMed]
- Sartor, R.B. Therapeutic correction of bacterial dysbiosis discovered by molecular techniques. Proc. Natl. Acad. Sci. USA2008, 105, 16413–16414. [Google Scholar] [CrossRef] [PubMed]
- Marchesi, J.R.; Holmes, E.; Khan, F.; Kochhar, S.; Scanlan, P.; Shanahan, F.; Wilson, I.D.; Wang, Y. Rapid and noninvasive metabonomic characterization of inflammatory bowel disease. J. Proteome Res.2007, 6, 546–551. [Google Scholar] [CrossRef] [PubMed]
Будьте здоровы!
ССЫЛКИ К РАЗДЕЛУ О ПРЕПАРАТАХ ПРОБИОТИКАХ
Вне моментов обострения диета при болезни Крона должна состоять из продуктов, которые были перечислены в начале статьи. Используя их, можно составить подобное дневное меню.
Диета при болезни Крона: меню на неделю
Если подытожить все, что было описано выше, мы получим конкретные рекомендации, которые касаются недельного рациона питания при подобном кишечном недуге. Кстати, примерно такую же диету рекомендуют пациентам, перенесшим операцию, ведь в этот период организму нужно дать время восстановиться.
Что же, разделим нашу диету на этапы, которые в общей сложности займут у нас неделю:
- Первые два дня – голодание. Можно пить чай, обезжиренный кефир, а в случаях поноса разрешено употреблять морковку и яблоки.
- Этап второй – протертые супы с мясом и легкие бульоны, сваренные на курином мясе. В меню можно добавить сухарики, кисель, паровой омлет и каши на воде. Данные ограничения действуют в течение 3 дней.
- На третьем этапе допускается употребление в пищу тушеных овощей. Также можно есть запеченные яблоки, сыры и нежирные молочные продукты. Разрешено вареное или приготовленное на пару мясо (курица или баранина), яйца, сваренные вкрутую и мелкая вермишель.
Разумеется, более точные наставления пациенты получают от лечащего врача. Диета должна составляться индивидуально.
Источники
- https://vrbiz.ru/raznoe/pitanie-krona
- https://customs.news/diets/kakaya-dieta-naznachaetsya-pri-bolezni-krona-chto-mozhno-i-nelzya-kushat-menyu-na-nedelyu/
- http://propionix.ru/mikrobiom-metabolom-i-vospalitelnoye-zabolevaniye-kishechnika
- https://autogear.ru/article/310/860/dieta-pri-bolezni-krona-menyu-i-osobennosti-pitaniya/